EMSIG - Ereignisgesteuerte MikroSkopie für die Intelligente mikrofluidische sinGle-Zell-Analyse
Gefördert durch die Helmholtz Imaging Platform
Contact: Erenus Yildiz, Hanno Scharr
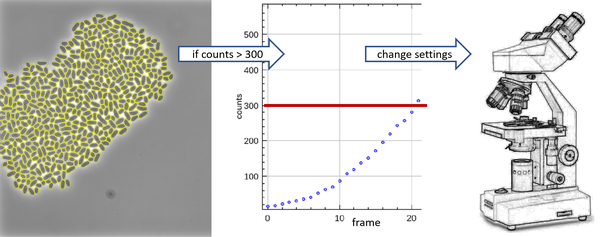
Die mikrofluidische Lebendzellanalyse bietet Bio(techno)logikern einzigartige Einblicke in die Wachstumsvielfalt und Populationsheterogenität einzelner mikrobieller Zellfabriken. Kürzlich entwickelte Deep-Learning-basierte Bildanalysemethoden und Tools aus dem SATOMI-Projekt bieten leistungsstarke Werkzeuge für eine Offline-Analyse hochwertiger Daten. Im Gegensatz zur Durchflusszytometrie, die sich vor allem als praktisches Werkzeug für das Einzelzell-Screening bewährt hat, steht die mikrofluidische Lebendzelltechnologie vor der Herausforderung, über Tage und Wochen hinweg qualitativ hochwertige Datensätze zu erfassen.
Das EMSIG-Projekt zielt darauf ab, unüberwachte Ereignisdetektion (ED) in die mikrofluidische Lebendzellanalyse einzubringen, die in der Lage ist, vordefinierte Ereignisauslöser aus Lebendzellmikroskopiedaten zu erkennen, diese Auslöser in Ereignistypen zu klassifizieren und die Ergebnisse mit einer computerisierten Steuerung der mikroskopischen Bildgebungskomponenten zu verbinden. Dies wird es ermöglichen, nicht nur die allmähliche Verschlechterung der Bildqualität, sondern auch biologische Ereignisse von Interesse live zu erkennen.
In einer ersten Phase werden wir ED in einem menschlichen Assistenzsystem realisieren, während in der zweiten Phase eine automatisierte Steuerung implementiert wird, um einer Verschlechterung der Bildqualität autonom entgegenzuwirken und sich auf spezifische biologische Ereignisse in einer erhöhten räumlichen und zeitlichen Auflösung zu konzentrieren.
Der ausgeprägte Hochrisikofaktor der zweiten Phase liegt in der robusten und genauen Klassifizierung der Ereignisauslöser an der Schnittstelle zwischen bildgebenden und stochastischen biologischen Komponenten, von der wir im Erfolgsfall hohe Erträge erwarten, da sie den Informationsgewinn pro Versuchszeit steigert, völlig neue Erkenntnisse zur Charakterisierung schneller Prozesse ermöglicht, den Automatisierungsgrad der Lebendzellmikroskopie erhöht und den Technology Readiness Level (TRL) der mikrofluidischen Lebendzellmikroskopie steigert.
Kooperationspartner:
Katharina Nöh, Forschungszentrum Jülich, IBG‐1: Biotechnology
Dietrich Kohlheyer, Forschungszentrum Jülich, IBG‐1: Biotechnology
Ralf Mikut, Karlsruhe Institute of Technology (KIT), Institute for Automation and Applied Informatics (IAI)
