„BrainBuilder“ macht 3D-Rekonstruktion des Gehirns aus 2D-Schnitten möglich
„BrainBuilder“ ist der Name einer neuen, KI-gestützten Rekonstruktions-methode, die die zahlreichen technischen Probleme bei der Umwandlung von 2D-Gehirnschnitten in 3D-Atlanten löst. Sie erlaubt die Erstellung detaillierter, hochauflösender Mesoskalen-Atlanten, insbesondere von Neurotransmitter-Rezeptoren – ein entscheidender Schritt, um die funktionelle Organisation des Gehirns besser zu verstehen. An der Entwicklung von „BrainBuilder“ war ein internationales Team von Forschenden beteiligt. Ihre Studie ist jetzt im Fachmagazin Communications biology erschienen.
Zu den Mesoskalen-Strukturen des Gehirns gehören die Schichten der Großhirnrinde und ihre Bausteine, sogenannte kortikale Säulen. Diese Strukturen lassen sich durch post-mortem 2D-Aufnahmen, zum Beispiel histologische Färbungen oder Rezeptor-Autoradiographie, hochauflösend darstellen. Besonders interessant ist dabei die Verteilung von Neurotransmitter-Rezeptoren, die eine zentrale Rolle für Informationsverarbeitung und Signalübertragung spielen, und die in den Rezeptor-Autoradiographien visualisiert wird. Mesoskalen-Atlanten spielen eine zentrale Rolle bei der Aufklärung der komplexen Architektur des Gehirns und geben Aufschluss über feine Details seiner Struktur, die wesentlich sind, um unser Verständnis von normaler Gehirnfunktion und pathologischen Veränderungen voranzubringen.
Bislang war eine 3D-Rekonstruktion von Rezeptor-Autoradiographien aus den 2D-Schnitten, sogenannten „Sections“, unmöglich. Eine einfaches „Stapeln“ der Schnitte ergibt meist kein korrektes 3D-Bild, da sie in unterschiedlichen Winkeln und Abständen vorliegen, teilweise Schnitte fehlen, deformiert sind oder mit unterschiedlichen Verfahren aufgenommen wurden. Ziel des Forschungsteams aus Jülich, Düsseldorf, London, Montreal und New York war es deshalb, eine (automatisierte) Technik für eine präzise 3D-Rekonstruktion zu entwickeln.
Ergebnis ist die in der Studie vorgestellte Pipeline „BrainBuilder“ – also ein Software-Werkzeug, das verschiedene Schritte der Bildverarbeitung und Analyse kombiniert und dieses so aufeinander abstimmt, dass am Ende ein nutzbares Gehirnmodell entsteht. KI spielt dabei auch mit: Basis ist ein Deep-Learning-Netzwerk (U-Net), das auf synthetischen Daten trainiert wurde. „Brainbuilder“ ist flexibel einsetzbar: bei unterschiedlichen Datentypen, bei Lücken zwischen den Schnitten, bei verschiedenen Gewebearten, ohne exakt passendes Vergleichsbild vom Spendergehirn und speziesübergreifend.
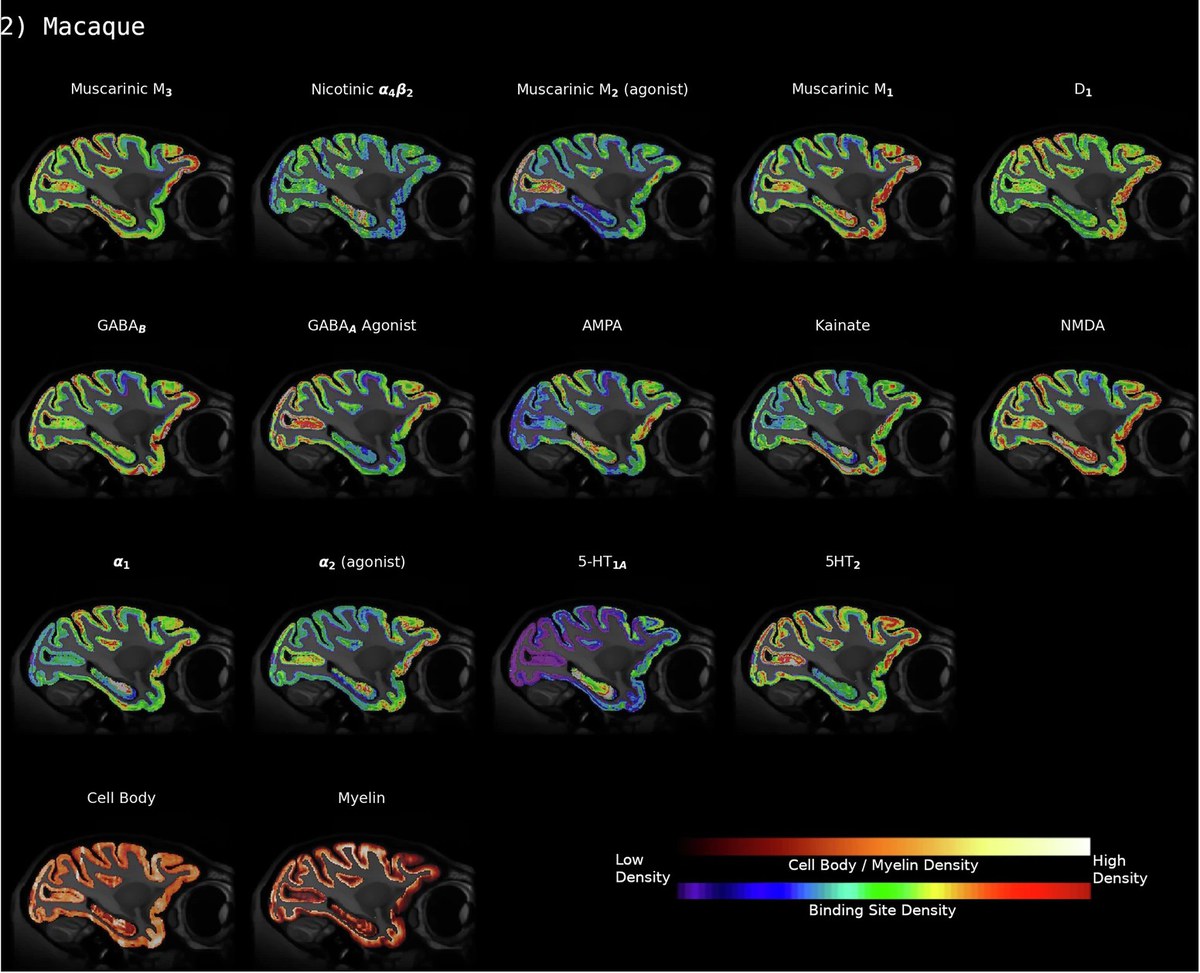
Zur Validierung testete das Team die Pipeline an zwei groß angelegten Datensätzen aus der Auswertung von Hirnschnitten: einem, der das gesamte menschliche Gehirn umfasst, und einem aus dem Gehirn eines Makaken. Sie wurden über mehr als zwanzig Jahre von den Neurowissenschaftlern Karl Zilles und Nicola Palomero-Gallagher an der Heinrich-Heine-Universität Düsseldorf und dem Forschungszentrum Jülich gesammelt und zeigen unter anderem detailliert die Verteilung verschiedener Rezeptoren der „klassischen“ Botenstoffe Glutamat, GABA, Acetylcholin, Noradrenalin, Serotonin, Dopamin und Adenosin.
Mithilfe von „Brainbuilder“ rekonstruierten die Forschenden erfolgreich aus dem vollständigen Datensatz Volumen und Verteilung von 20 Rezeptoren in einer Hälfte des menschlichen Gehirns; in dem Makaken-Gehirn war es eine Auswahl von 14 Rezeptoren, dazu kam hier die Rekonstruktion von Zellkörpern und Myelin-gefärbten Schnitten. Für das menschliche Gehirn stand eine MRT-Aufnahme als Referenz zur Verfügung, nicht aber für das des Makaken.
Als ein wichtiges Ergebnis sehen die Wissenschaftler:innen in „Brainbuilder“ die Möglichkeit, eine Vielzahl von 2D-Datensammlungen des Gehirns im Mesoskalen-Bereich dreidimensional zu rekonstruieren. Dadurch eröffnen sich für die Neurowissenschaften neue Möglichkeiten, das Gehirn in bestimmten Bereichen noch detailreicher abzubilden und damit über verschiedene Spezies hinweg zu verstehen und zu vergleichen.
Originalpublikation:
Funck, T., Wagstyl, K., Lepage, C. Palomero-Gallagher, N. et al. Brainbuilder: a software pipeline for 3D reconstruction of cortical maps from multi-modal 2D data sets. Commun Biol 8, 1015 (2025). https://doi.org/10.1038/s42003-025-08267-6
Kontakt
apl-.Prof. Dr. rer. nat. Nicola Palomero-Gallagher
Working Group Leader "Receptors"
- Institut für Neurowissenschaften und Medizin (INM)
- Strukturelle und funktionelle Organisation des Gehirns (INM-1)
Raum 2005b
Pressekontakt
Erhard Zeiss
Wissenschaftlicher Kommunikationsreferent
- Institut für Neurowissenschaften und Medizin (INM)
- Strukturelle und funktionelle Organisation des Gehirns (INM-1)
Raum 3033


