JUWELS im Einsatz: Forschende entwickeln mit Supercomputer KI-Modell zur Vorhersage von Enzymfunktionen
Ein interdisziplinäres Team von Experten aus dem Forschungszentrum Jülich, der HHU Düsseldorf und der Helmholtz AI am Helmholtz-Zentrum München hat ein neues Modell für maschinelles Lernen auf molekularer Ebene entwickelt. Das KI-Modell „TopEC“ analysiert Enzymstrukturen, erlernt deren chemische Reaktionen und kann davon ihre Funktionen ableiten. Ein großer Schritt für die Enzymtechnik und Biokatalyse. Trainiert wurde das Model auf dem JUWELS Supercomputer des JSC.

Proteine bilden die Grundlage allen zellulären Lebens. Die Kenntnis von ihrer dreidimensionalen Struktur, die bereits 1958 erkannt wurde, bestimmt seither den Fortschritt in der Molekularbiologie, Medizin und Biotechnologie.
Die Form eines Proteins wird durch die Wechselwirkungen seiner Atome bestimmt. Es ist diese Struktur, die vorgibt, wie das Protein durch die Interaktion mit anderen Molekülen funktioniert. Fortschritte in der Vorhersage von Proteinstrukturen machen es insbesondere seit der Einführung von AlphaFold möglich, die Strukturen von Enzymen genau nachzubauen. AlphaFold ist ein KI-Programm, das Neuronale Netzwerke nutzt, um die dreidimensionale Struktur von Proteinen auf Basis ihrer Aminosäuresequenz vorherzusagen. Ein wichtiger Schritt, denn es braucht eine präzise Prognose von Enzymfunktionen, um nachhaltige, biobasierte Prozesse zu entwickeln und Genomdaten genau zu interpretieren.
Neues Modell für maschinelles Lernen erkennt Enzymfunktionen
Umfangreiche Datenbanken sind mit Strukturmodellen befüllt. Dennoch liegt bisher nur für etwa 60 Prozent aller bekannten Enzymfunktionen ein solches vor. Um diese Lücke zu schließen, hatte Gohlke bereits vor zwei Jahren mit einer Gruppe von Wissenschaftlern des Instituts für Bio- und Geowissenshaften der HHU Düsseldorf im Rahmen einer Forschungsarbeit die Datenbank „TopEnzyme“ entickelt. (DOI: 10.1093/bioinformatics/btad116). Jetzt soll darin nach und nach ergänzt werden, was bislang noch fehlt.
Dieses Forschungsteam um Prof. Gohlke hat nun gemeinsam mit KI-Expert:innen von Helmholtz AI (Helmholtz München) das neue Modell für maschinelles Lernen entwickelt: „TopEC“ beurteilt Enzymfunktionen auf der Grundlage von mehr als 250.000 Strukturen aus der Protein- und AlphaFold-Datenbank. Trainiert wurde das Modell auf dem JSC Supercomputer JUWELS.
Rechnergestützte Methoden sollen Datenlücken schließen
Eine Datenbank bereitzustellen, ist das eine – sie mit Daten zu füttern, das andere. Denn die molekularen Funktionen eines Enzyms auf der Basis ihrer vorhergesagten Struktur präzise zu bestimmen, bleibt eine Herausforderung. Die Funktion eines Enzyms lässt sich nicht immer 1:1 von seiner Struktur ableiten – das macht die das experimentelle Bestimmen von Enzymfunktionen nicht nur zeitaufwändig, sondern auch fehleranfällig. So überrascht es nicht, dass in vorhandenen Datenbanken mitunter inkorrekte Funktionszuweisungen enthalten sind.
Abhilfe schaffen können rechnergestützte Methoden, die direkt auf der Enzymstruktur basieren. Indem sie Funktionsvorhersagen in großem Umfang automatisiert treffen, können sie Datenlücken schnell und fehlerfrei schließen – und damit einen elementaren Beitrag auch zur korrekten Auswertung biologischer Daten liefern.
Entwicklung und Training auf Supercomputer JUWELS
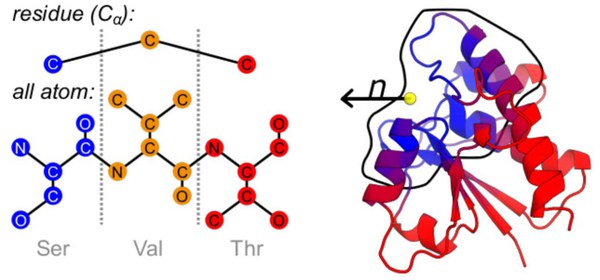
Um das Modell zu entwickeln und zu trainieren nutzte das Team Rechenzeit auf dem Supercomputer JUWELS am JSC, bereitgestellt durch das John von Neumann-Institut für Computing (NIC). Dabei konnten sie die Rechenanforderungen durch ein besonderes Vorgehen senken: Statt die vollständige Enzymstruktur zu verwenden, implementierten die Forschenden einen lokalisierten, atom-basierten 3-D-Deskriptor, der sich auf die nächsten hundert Atome um das aktive Zentrum eines Enzyms konzentrierte. Die Trainingsgeschwindigkeit stieg signifikant an.
Indem TopEC aus der Enzymstruktur weitere Informationen – etwa Abstände und Winkel zwischen Atomen – einbezieht, steigt die Genauigkeit in der Vorhersage von Enzymfunktionen im Vergleich zu herkömmlichen Methoden deutlich. Zudem ist das Modell besonders robust gegenüber strukturellen Variationen in Enzymbindungsstellen und kann ähnliche Funktionen über verschiedene Strukturmerkmale hinweg erkennen.
Einen möglichen Einsatzzweck stellt die gezielte Suche nach neuen Enzymen dar. Mit TopEC lassen sich rein rechnergestützt neue Enzymvarianten identifizieren. Das bietet insbesondere im Kontext der nachhaltigen Biotechnologie ganz neue Möglichkeiten.
Die Herausforderung, vor der sich die Wissenschaftler:innen nun gestellt sehen, besteht vor allem darin, dass es bereits mehr als 30 Millionen Enzyme mit vorhergesagten Funktionen gibt – meist basierend auf Sequenzvergleichen. Die tatsächliche Fehlerquote dieser Vorhersagen ist nicht genau bekannt. Diese Daten zu verfeinern, könnte die erste große Aufgabe für TopEC sein, und zwar auf Basis möglichst vieler automatisch generierter Strukturmodelle – etwa durch AlphaFold, einem KI-Programm, das Neuronale Netzwerke nutzt, um die dreidimensionale Struktur von Proteinen auf Basis ihrer Aminosäuresequenz vorherzusagen. Das Potenzial dieser Methode wollen Gohlke und sein Team nun in einem Folgeprojekt untersuchen.
Helmholtz School for Data Science in Life, Energy, and Earth (HDS-LEE) / Helmholtz AI
Die HDS-LEE Graduate School ist eine internationale englischsprachige Graduiertenschule, die sich an exzellente Graduierte in Mathematik, Informatik, Naturwissenschaften und Ingenieurwesen richtet, die die Entwicklung datenwissenschaftlicher Methoden verbessern und modernste datenwissenschaftliche Technologien zur Lösung anspruchsvoller wissenschaftlicher Probleme einsetzen wollen.
Helmholtz AI ist eine anwendungsorientierte Plattform für künstliche Intelligenz, die die Wissenschaft in der gesamten Helmholtz-Gemeinschaft beschleunigt. Sie ermöglicht die Entwicklung und Umsetzung von KI-Lösungen, fördert die Zusammenarbeit und stellt den Zugang zu Ressourcen und Fachwissen sicher.
Originalpublication: TopEC: prediction of Enzyme Commission classes by 3D graph neural networks and localized 3D protein descriptor, by van der Weg, K., Merdivan, E., Piraud, M., Gohlke, H. Nature Commun. 2025, 16, 2737. DOI: 10.1038/s41467-025-57324-5
Kontakt: Prof. Dr. Holger Gohlke
Weitere Informationen:
HDSLEE: HDS-LEE - Helmholtz Information & Data Science Academy
Helmholtz AI: Helmholtz AI
Bioinformatik am FZJ: Bioinformatik (IBG-4)
