Strukturelle und funktionelle Organisation des Gehirns (INM-1)
Im INM-1 wird ein 3D-Modell des menschlichen Gehirns entwickelt, das Zytoarchitektonik, Konnektivität, molekulare Gliederung sowie Genetik und Hirnfunktion berücksichtigt.
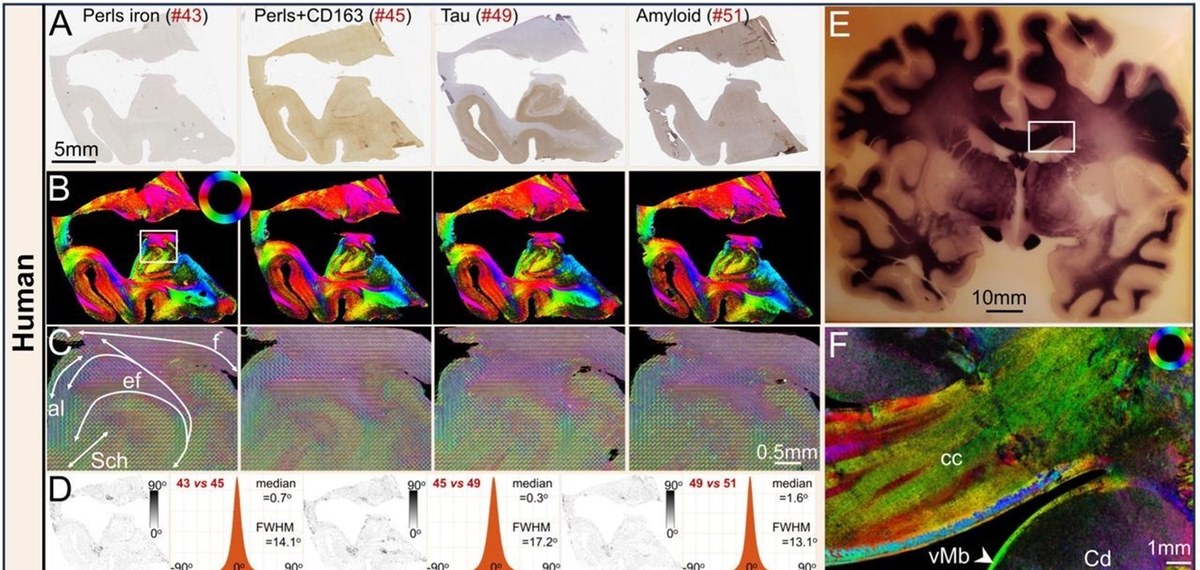
Einem internationalen Team von Wissenschaftler:innen ist es gelungen, die kürzlich entwickelte Bildgebungstechnik ComSLI erstmals auf ganz unterschiedlich präparierten Gehirnschnitten anzuwenden und damit das komplexe Fasernetzwerk des Gehirns mit Mikrometer-Genauigkeit abzubilden. Das ist ein Fortschritt, der der neurowissenschaftlichen und biomedizinischen Forschung neue Möglichkeiten eröffnet – zum Beispiel auch einer erneuten, vertieften Untersuchung bereits vorhandener Gewebeschnitte. Ihre Untersuchung ist jetzt in der renommierten Fachzeitschrift Nature Communications erschienen.
Eine genaue Kartierung der Nervenfasern im Gehirn ist wichtig, um seine Funktionen und mögliche Erkrankungen besser verstehen zu können. Allerdings ist es schwierig, das komplexe Netzwerk von Nervenzellen über große Bereiche und mit hoher Genauigkeit abzubilden. Die Streulicht-Technik ComSLI ist eine Methode, mit der die Richtung und Verläufe von komplexen Faserstrukturen im Gehirn mit Mikrometer-Auflösung sichtbar gemacht werden. Dabei wird ein hauchdünner Gehirnschnitt aus verschiedenen Richtungen mit Licht durchleuchtet und das senkrecht gestreute Licht mit einer hochauflösenden Kamera gemessen. Das gestreute Licht gibt Aufschluss darüber, wie die Fasern angeordnet sind und wie sie sich kreuzen.
Die Forschenden aus Stanford, Delft, Rotterdam und aus dem Institut für Neurowissenschaften und Medizin (INM-1) in Jülich konnten den Anwendungsbereich von ComSLI wesentlich erweitern. In ihrer Studie demonstrieren sie, wie die neue Technik die Netzwerke von Faserbahnen in unterschiedlichen histologischen Präparaten, unabhängig von deren Präparierung, mikrometergenau abbilden kann, z.B. in zellkörpergefärbten Ganzhirnschnitten des Gehirns, bei unterschiedlichen Einbettungsverfahren und bei ungefärbten oder frischen Gewebeschnitten. Dies war bislang nicht möglich. Ein Teil der Schnitte der Studie stammt aus dem Institut für Neurowissenschaften und Medizin (INM-1) in Jülich sowie dem Cécile und Oskar Vogt-Institut für Hirnforschung in Düsseldorf. Die Studie baut auf dem Wissen und der Erfahrungen auf dem Gebiet der 3D-Polarisationsbildgebungauf, mit der erstmals die feine Faserbahnarchitektur in Präparaten des Gehirns in ihrem dreidimensionalen Verlauf sichtbar gemacht werden konnte.
Die neue Methode eröffnet nun die Möglichkeit, aus bereits bestehenden Schnittserien durch ComSLI neue, zusätzliche Informationen zu gewinnen. So wurden in einem Schnitt eines zweiten „BigBrain“ Datensatzes die Faserverläufe zusätzlich zur bereits bestehenden Zellkörperverteilung visualisiert. Dieses zweite „BigBrain“ ist ähnlich wie sein Vorgänger, der Kernstück der europäischen Forschungsinfrastruktur EBRAINS ist, eine dreidimensionale Rekonstruktion eines ganzen menschlichen Gehirns, basierend auf mehreren Tausend zellkörpergefärbten Serienschnitten.
Der Ansatz verspricht auch neue Erkenntnisse in der medizinischen Diagnostik: Die Wissenschaftler:innen konnten verändertes Gewebe bei Erkrankungen wie Multipler Sklerose und Leukenzephalopathie erkennen, geschädigte Nervenbahnen zum Beispiel bei Schädigungen des Hippocampus und bei Alzheimer sichtbar machen und zeigen, dass dieses Verfahren Vorteile gegenüber bisherigen Untersuchungsmethoden bietet.
In ihrer Studie gingen die Forschenden noch einen Schritt weiter. Neben Hirngewebe kartierten sie auch Fasern in anderen Gewebearten wie Muskeln, Knochen und Blutgefäßen. Auch hier wurden feinste Faserstrukturen sichtbar gemacht, die Rückschlüsse auf ihre Funktion liefern. Damit wird deutlich, dass die Methode nicht nur für die Erforschung des Gehirns, sondern auch anderer Organe und Gewebetypten großes Potenzial hat und beitragen kann, pathologische Prozesse aufzuschlüsseln.
Georgiadis M, von der Heiden F, Abbasi H, Ettema L, Nirschl J, Taghavi HM, Wakatsuki M, Liu A, Ho WHD, Carlson M, Doukas M, Koppes SA, Keereweer S, Sobel RA, Setsompop K, Liao C, Amunts K, Axer M, Zeineh M, Menzel M. Micron-resolution fiber mapping in histology independent of sample preparation. Nat Commun 16, 9572 (2025). https://doi.org/10.1038/s41467-025-64896-9
Working Group Leader "Fiber Architecture" Deputy Head of INM-1
Director of the INM-1 and Working Group Leader "Architecture and Brain Function"
Assistant Professor, TU Delft
Wissenschaftlicher Kommunikationsreferent